
電話:
021-67610176傳真:
在一項新的研究中,一項突破性的新技術使得科學家們一次能夠成像觀察單個細胞內的10421個基因。這項研究是在美國加州理工學院神經科學研究所生物學研究教授Long Cai的實驗室中完成的。相關研究結果于2018年6月7日在線發表在Cell期刊上,論文標題為“Dynamics and Spatial Genomics of the Nascent Transcriptome by Intron seqFISH”。
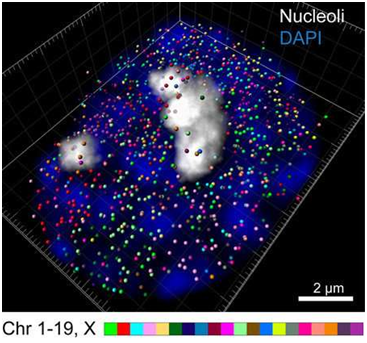
圖片來自Cai laboratory。
這種被稱為內含子seqFISH(sequential fluorescence in situ hybridization, 連續熒光原位雜交)的新技術是在能夠一次識別上百個細胞的基因組中發生什么上取得的一項重大進展。在此之前,人們僅能夠利用顯微鏡一次對細胞中的4到5個基因進行成像觀察。這項研究建立在Cai實驗室取得的早前進展的基礎之上,包括2014年的seqFISH早期版本和2017年在顯微鏡下對1萬多個基因進行分析的研究。如今,將seqFISH的規模擴大到基因組水平使得能夠對單個細胞內的10000多個基因---大約占哺乳動物基因總數的一半---進行成像觀察。
為了將基因中的遺傳指令轉化為具有實際功能的蛋白,一種被稱作轉錄的過程首先必須發生。這個過程經常以脈沖或“突發”的形式發生。首先,基因經轉錄后產生信使RNA前體(pre-mRNA)。pre-mRNA隨后經剪接后產生成熟的信使RNA(mRNA)。在這種剪接過程中,某些被稱為內含子的區域從pre-mRNA中切除。
Cai團隊選擇著重關注對內含子進行標記,這是因為它們是在轉錄過程的早期產生的,這有助了解細胞在基因表達的時刻在做什么。
利用這種新開發的內含子seqFISH技術,每個內含子都標記上一種*的熒光條形碼,這就是使得能夠利用顯微鏡進行觀察。對內含子進行觀察揭示出哪些基因當前在單個細胞中表達,它們的表達強度和它們所在的位置。這樣一次就能夠對10421個內含子---因而對10421個基因---進行成像觀察。
之前開發條形碼技術的研究工作主要集中在對mRNA本身進行標記,從而測量在mRNA產生后的幾個小時內基因表達如何發生變化。研究內含子使得這些研究人員研究所謂的新生轉錄組(nascent transcriptome)---新合成基因的表達。這導致他們發現相比于細胞發生分裂和進行自我復制所需的時間(大約12~24個小時),基因轉錄在“非常短”的時間范圍內(僅大約兩個小時)在許多基因上發生全局性地波動。這意味著在兩個小時的時間內,細胞內的許多基因將會突然地開啟和關閉。
之前沒有觀察到這種波動現象有幾個原因。首先,鑒于這兩個小時內的波動在不同細胞之間并不同步,因此這些波動在需要很多細胞的方法中被平均掉。其次,這種seqFISH方法的高度準確性允許這些研究人員能夠確定他們觀察到的是真實的生物波動,而不是技術噪音。后,當測量的對象是mRNA而不是內含子時,這兩個小時內的波動被掩蓋掉了,這是因為mRNA分子在哺乳動物細胞中具有較長的壽命(3~4個小時)。
此外,鑒于內含子停留在基因所在的物理位置上,因此對內含子進行熒光成像觀察允許人們能夠可視化觀察基因在染色體中的位置。在這項研究中,Cai團隊吃驚地發現大多數有活性的蛋白編碼基因位于染色體表面上,而不是埋藏在它的內部。
Cai說,“這種技術能夠應用于任何組織。除了讓我們觀察相同細胞中的染色體結構之外,內含子seqFISH能夠有助于識別細胞類型,以及細胞要做什么。”(生物谷 )
參考資料:
Sheel Shah6, Yodai Takei6, Wen Zhou et al. Dynamics and Spatial Genomics of the Nascent Transcriptome by Intron seqFISH. Cell, Published online: June 7, 2018, doi:10.1016/j.cell.2018.05.035