
電話:
021-67610176傳真:
細胞通過基因組復制產生自身的拷貝而進行增殖。按理說,DNA復制是所有生命形式中基本和保守的機制。破解這一過程是如何地實現的秘密是理解生命秘密的關鍵。當沃森和克里克在半個多世紀前基于DNA雙螺旋結構提出DNA的復制方式時,許多人認為將兩條DNA鏈分開進行復制的分子機器(即DNA復制機器,或者說DNA復制復合物)的結構即將出現。然而,鑒于這種分子機器具有比較大的尺寸、三重特性(它由三個引擎組成)和靈活性,它遠要比之前想象的復雜得多。利用常規方法無法獲得這種DNA復制機器在原子分辨率下的結構信息。近年來,隨著高分辨率的低溫電鏡(cryo-EM)技術的來臨,人們才能獲得它在原子分辨率下的結構信息。
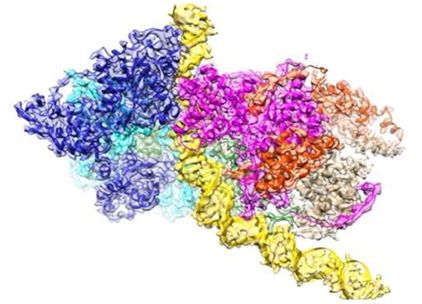
圖片來自Division of Life Science, The Hong Kong University of Science and Technology。
中國北京大學的高寧(Ning Gao)團隊和中國香港科技大學的Bik-Kwoon Tye團隊合作發表的一系列論文為以的分辨率破解DNA復制機器的功能打開了大門。篇論文于2015年發表在Nature期刊上,它解析出這種被稱作微小染色體維持蛋白復合物(minichromosome maintenance complex, MCM蛋白復合物)的DNA復制機器的核心引擎的結構。第二篇論文報道了Cdt1-Mcm2-7復合物(作為MCM雙六聚體復合物的前體)的開環結構。如今,第三篇論文發表在Nature期刊上,它詳細地說明了起始識別復合物(Origin Recognition Complex, ORC)的原子結構,其中ORC復合物選擇全基因組中的復制起始位點以便啟動DNA復制。
在單個細胞(受精卵)經過大約1016個細胞分裂后,每個人就產生了。每次細胞分裂都需要基因組的復制,這樣每個子細胞都以DNA的形式獲得*相同的遺傳信息。
導致細胞分裂失調的異常DNA復制是許多癌癥和發育障礙的原因。基因組復制在維持活的有機體方面同樣起著重要的作用,這是因為所有細胞都具有“有效期限”,并且大多數細胞都由保留分裂能力的干細胞進行補充。
衰老也是這種復制機器發生故障(無論是發生在受損DNA的修復合成中,還是發生在全基因組復制的保真度中)的一般現象。在第三篇論文中,中國北京大學的高寧教授和中國香港科技大學的Bik-Kwoon Tye教授和Yuanliang Zhai博士利用低溫電鏡技術在原子分辨率下解析出啟動DNA復制的ORC復合物的結構。這種結構解釋了ORC復合物如何能夠掃描大量的堿基(DNA由A、T、G和C四種堿基組成),從中選擇出正確的位點開始進行DNA復制。據信,不加區分地選擇太多的位點可能導致基因組的快速復制并因此導致快速的細胞分裂,這是癌細胞的特征。相比之下,低效地選擇位點會導致細胞分裂緩慢,特別是在人類發育的關鍵時刻,這可能導致發育障礙。
一個典型的病例是Meier-Gorlin綜合征(MGS),這是一種罕見的遺傳性侏儒癥,其特征是產前發育遲緩和產后比例矮小的身材。有趣的是,與Meier-Gorlin綜合征相關的突變位于5個基因(ORC1、ORC4、ORC6、CDT1和CDC6)中,所有的這些基因都是這種DNA復制起始機器(即ORC復合物)的組分。ORC1和ORC4發生突變的患者似乎具有嚴重的身材矮小。在第三篇論文中,高寧教授和Bik-Kwoon Tye教授等證實在形成這種DNA復制起始機器的六個亞基中,ORC1和ORC4在復制起始位點的選擇機制中起著決定性作用。
從根本上說,ORC復合物的重要功能是招募MCM雙六聚體復合物到DNA復制起點上,其中MCM雙六聚體復合物是將雙鏈DNA分離開的DNA解旋酶的催化核心。在第三篇論文中報道的ORC復合物結合到DNA上時的原子結構揭示出ORC復合物導致的DNA彎曲為DNA插入到MCM解旋酶的開環結構中提供一個停泊表面。再者,它揭示出DNA復制起點是基于它的*結構而不是基于它的特定堿基序列而被選擇出來的。這些新發現有助于解釋ORC復合物如何在基因組中的*位點上選擇復制起點,而且這些位點僅由它們的堿基序列是無法預測到的。
DNA復制是所有有機體的一個決定性特征,而且執行這一功能的DNA復制機器從真菌到植物到人類都是保守的。理解這種DNA復制機器(或任何生物分子機器)的原子結構是至關重要的,這是因為所有的應用技術和工程技術都建立在基礎科學/知識的基礎之上。比如, DNA復制機器在3埃分辨率下的三維結構圖可能有助我們鑒定出更好的癌癥治療靶標,這樣就可定制產生適合這種靶標的合成化學物。更重要的是,這些結構有助于我們充分理解分子機器的作用機制,從而有助我們理解因這些分子機器未發揮功能而導致的疾病的根源。為實現這一目標,中國香港科技大學將建立先進的低溫電子顯微設備,用于研究生物分子機器的高分辨率結構。(生物谷 )
參考資料:
1.Ningning Li et al. Structure of the eukaryotic MCM complex at 3.8 Å.Nature (2015), doi:10.1038/nature14685.
2.Yuanliang Zhai et al. Open-ringed structure of the Cdt1–Mcm2–7 complex as a precursor of the MCM double hexamer. Nature Structural & Molecular Biology (2017), doi:10.1038/nsmb.3374.
3.Ningning Li et al. Structure of the origin recognition complex bound to DNA replication origin. Nature (2018), doi:10.1038/s41586-018-0293-x.